Featured Scientist
Wei-Ning Lin
Wei-Ning Lin
Graduate Institution of Biomedical and Pharmaceutical Science,
Fu Jen Catholic University, New Taipei City, Taiwan
Article published in
"Journal of Clinical Medicine" 2018, 7(9), 266
代謝體研究於重症肌無力患者與腸道菌相改變
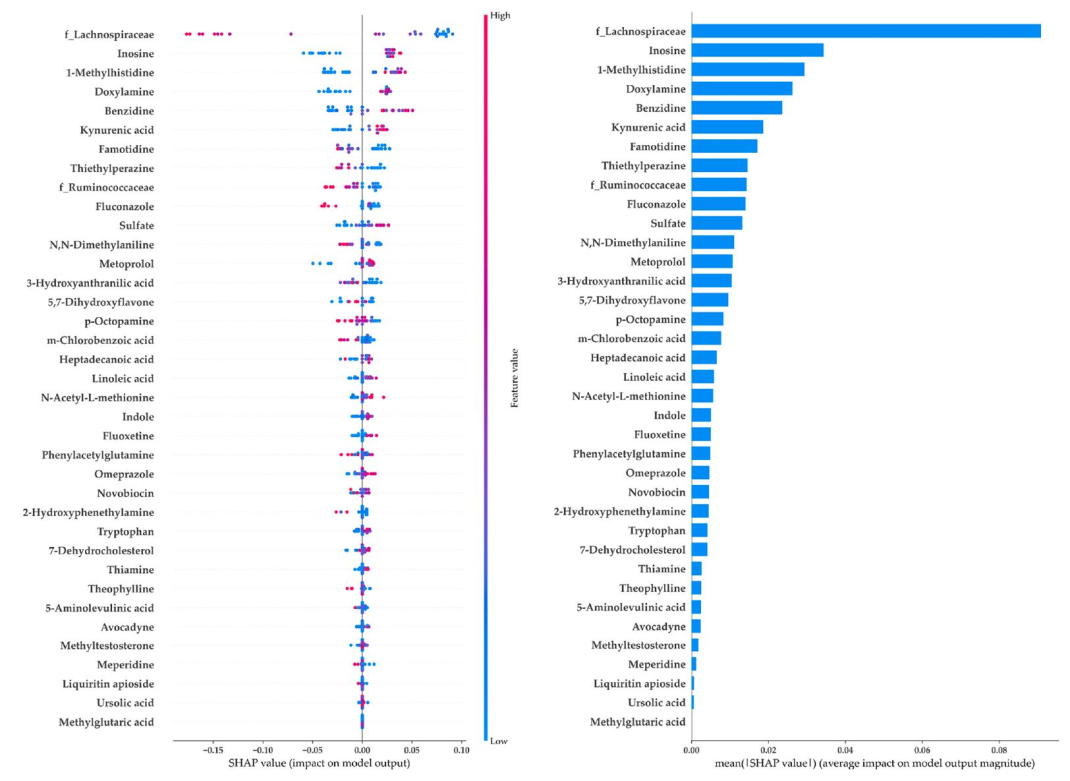
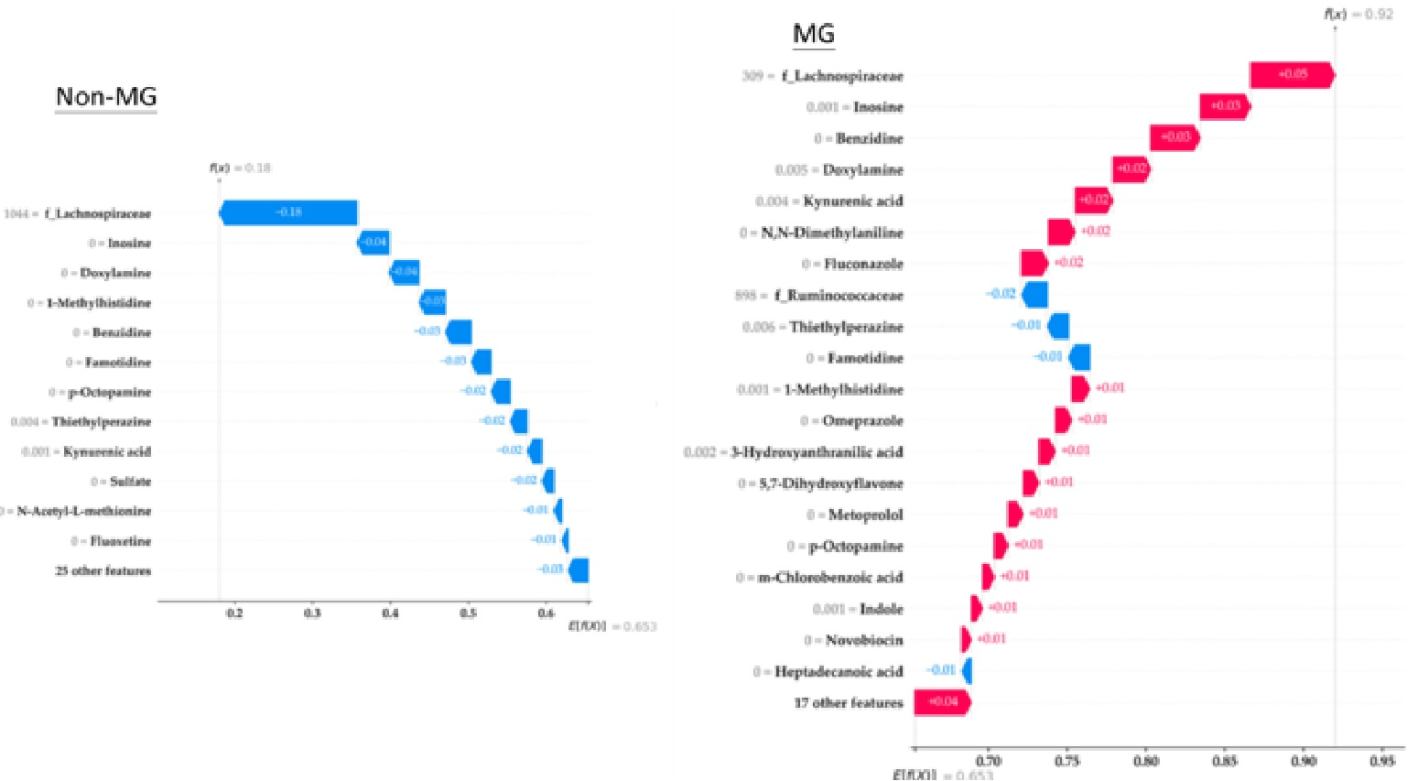
接續之前實驗,我們嘗試加入代謝物的分析來強化解析MG、腸道菌相與代謝物之間的關聯並進行機器學習(machine learning, ML)的開發。我們納入19名MG患者與10名非MG受試者,收集糞便樣本並以16S rRNA定序分析微生物組;同時進行非靶向代謝體分析,鑑定糞便中的擴增子序列顯著變異(amplicon significant variants, ASVs)及代謝物。研究建立的可解釋 ML模型整合排名前列的ASVs與代謝物,並透過 SHapley Additive exPlanations (SHAP)生成全域及個人化解釋。結果顯示,兩組在糞便微生物-代謝物組成上存在顯著差異,關鍵菌科為Lachnospiraceae與Ruminococcaceae,而前三大特徵依序為Lachnospiraceae、肌苷(inosine)及甲基組氨酸(methylhistidine)。以前1% ASVs結合前15%代謝物訓練的ML模型表現優於其他模型。個人化SHAP解釋顯示MG患者之微生物-代謝物貢獻模式各異。整合微生物與代謝物特徵並建立可解釋ML架構,可準確鑑別MG,並提供個別化解釋,揭示腸道菌群、代謝物與MG的關聯。此成果發表於Computational and Structural Biotechnology Journal期刊(2024)。
Keywords:重症肌無力(myasthenia gravis)、腸道菌相(gut microbiota)、 代謝物(metabolites)、機器學習(machine learning)
12 views